
ノックアウトマウスを作ってみたいけど、どこから手をつければいいの?

ガイドRNAの設計って、どうやってやるの?
ゲノム編集技術の進化により、遺伝子ノックアウト(KO)は以前よりも手軽に行えるようになりましたが、設計の段階でつまずいてしまう研究者も少なくありません。
本記事では、KOマウスの作製に必要な情報収集からガイドRNAの設計まで初心者でも分かるように解説します。特に、Hprt遺伝子を例にした具体的な設計ポイントを紹介することで、初めての方でも迷わず進められるように構成しています。
さらに、MGI、IMPC、KOnezumiなどの有用なウェブページや専門ツールの活用方法も詳しく紹介。これらを使いこなすことで、設計の精度と効率が格段に向上します。
この記事を読むことで、あなたは「どのエキソンを標的にすべきか」「どんなガイドRNAを設計すればよいか」といった疑問を解消し、目的に合ったKOマウスを自信を持って設計・作製できるようになります。
ゲノム編集の力を最大限に活かし、研究の質を高める一歩を踏み出しましょう!
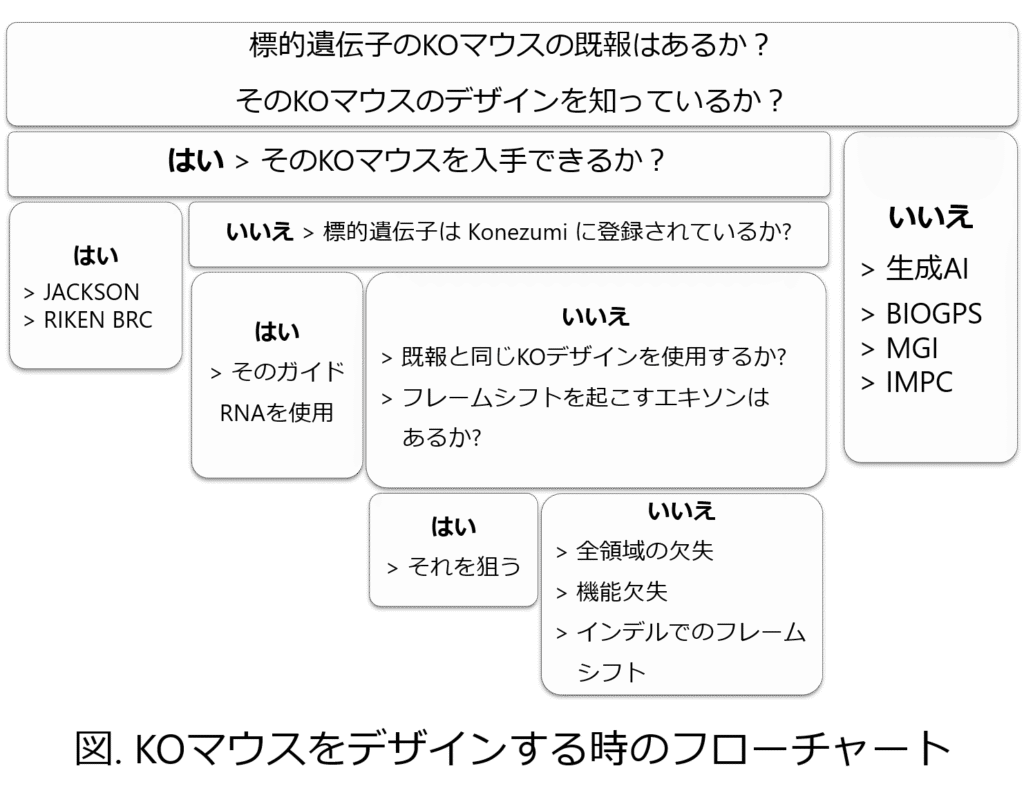
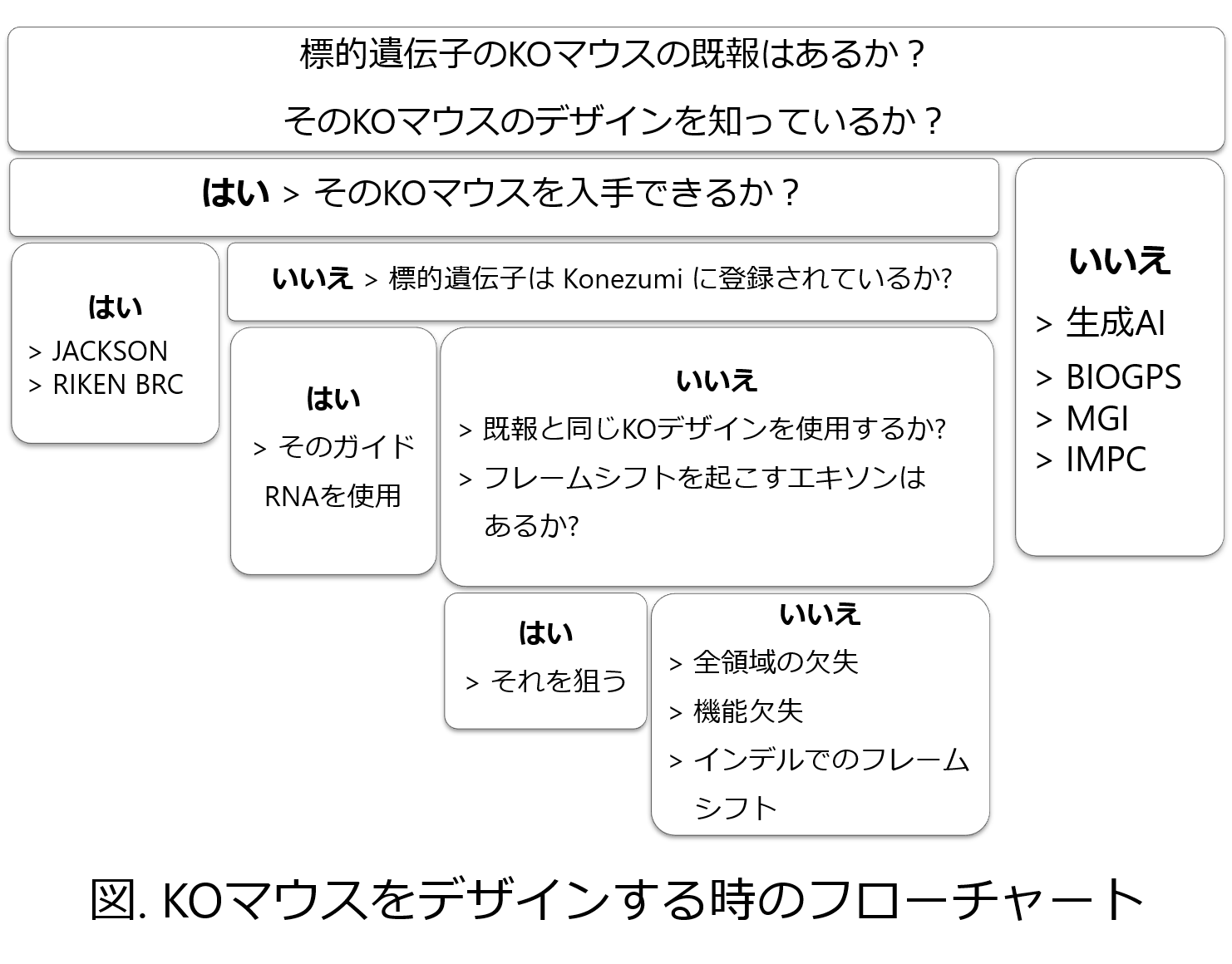
標的遺伝子のKOマウスの既報はあるか? そのKOマウスのデザインを知っているか?
本記事では、Hprt 遺伝子を例に取り、ガイドRNAの設計ポイントについて詳しく解説します。
まずは「標的遺伝子のKOマウスの既報はあるか(既報を知っているか)? そのKOマウスのデザイン(設計情報)を知っているか?」という問いからスタートしましょう。
いいえ:KOマウスの既報を調査(生成AI, BIOGPS, MGI, IMPC)
KOマウスに関する情報を持っていない(KOマウスについてよく知らない)場合は、まずは検索に強い生成AI「Perplexity」を活用してみましょう。
たとえば、「○○遺伝子のノックアウトマウスの表現型を教えてください」と入力するだけで、関連情報を得ることができます。ただし、ソースを確認して情報の真偽に注意を払いましょう。

研究における生成AIの利用については、下記の記事も参照してください。
もちろん、Google, PubMed, Google Scholarなどの検索エンジンやデータベースでKOマウスについて調べるのも有効です。
BioGPSはヒト・マウス・ラットの様々な組織や細胞における遺伝子発現データを包括的に提供するデータベースです。
このツールを使えば、各臓器・組織での遺伝子発現パターンを調べることができ、KOした場合に表現型が現れる可能性が高い臓器・組織を予測することもできるかもしれません。
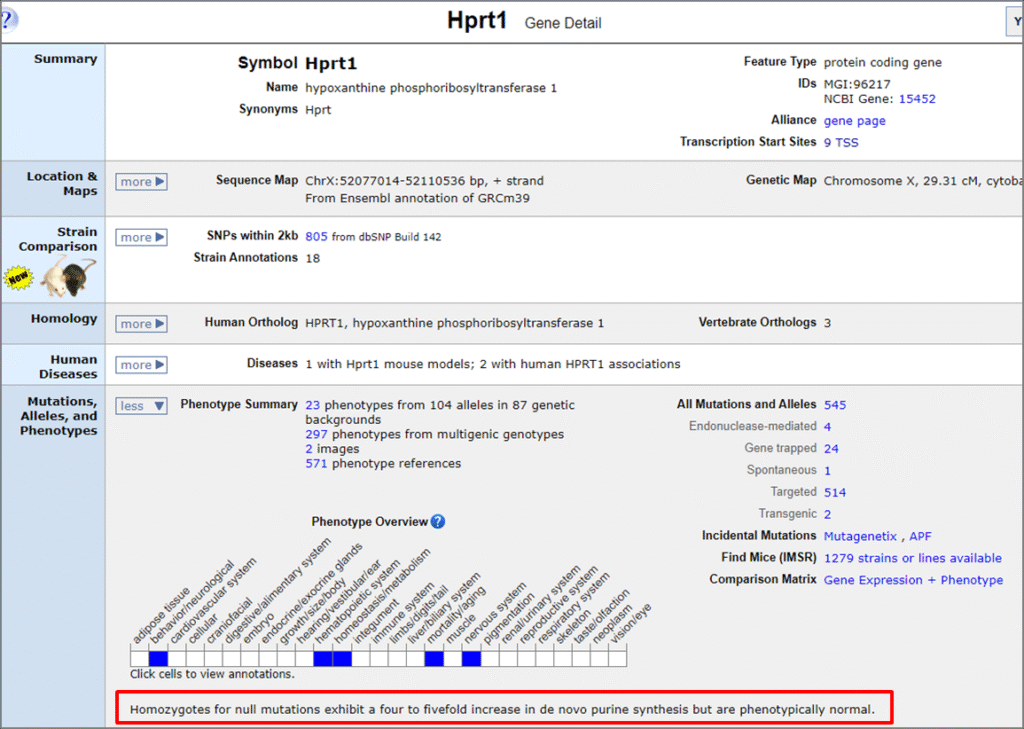
MGI (Mouse Genome Informatics) はマウス研究における最も包括的なデータベースの一つです。マウス遺伝子について調べる際に、多くの研究者がまず最初にアクセスするサイトかもしれません。
遺伝子名を入力するだけで、遺伝子の発現データ、KO表現型、関連文献など、研究に必要な多くの情報を一度に見ることができます。
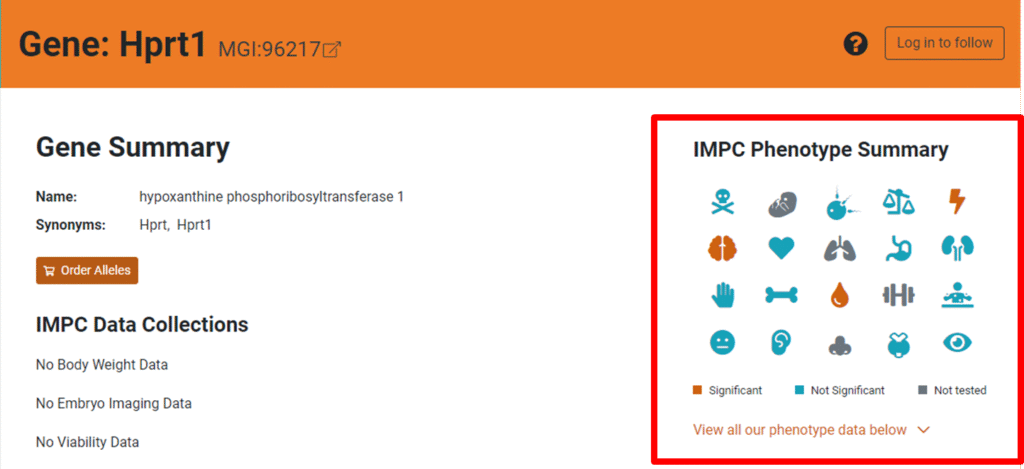
IMPC(International Mouse Phenotyping Consortium)は国際マウス表現型解析コンソーシアムの略称で、マウスの全遺伝子について体系的にKOマウスを作製し、標準化された手法で網羅的な表現型解析を行う大規模プロジェクトです。
IMPCのサイトでは、遺伝子KOの詳細な影響や表現型情報を入手できます。現状、すべてのマウス遺伝子の情報が網羅されているわけではありませんが、目的の遺伝子の情報が登録されていれば、有用な参考資料となります。
はい:そのKOマウスを入手できるか?
はい:市販のKOマウスの活用(Jackson Laboratory, RIKEN BRC)
KOマウスの表現型や既存研究について十分に理解したら、次に検討すべきは既存のKOマウスが入手可能かどうかです。場合によっては、自分で作製するよりも購入する方が早く、簡単で、コスト面でも有利になることがあります。
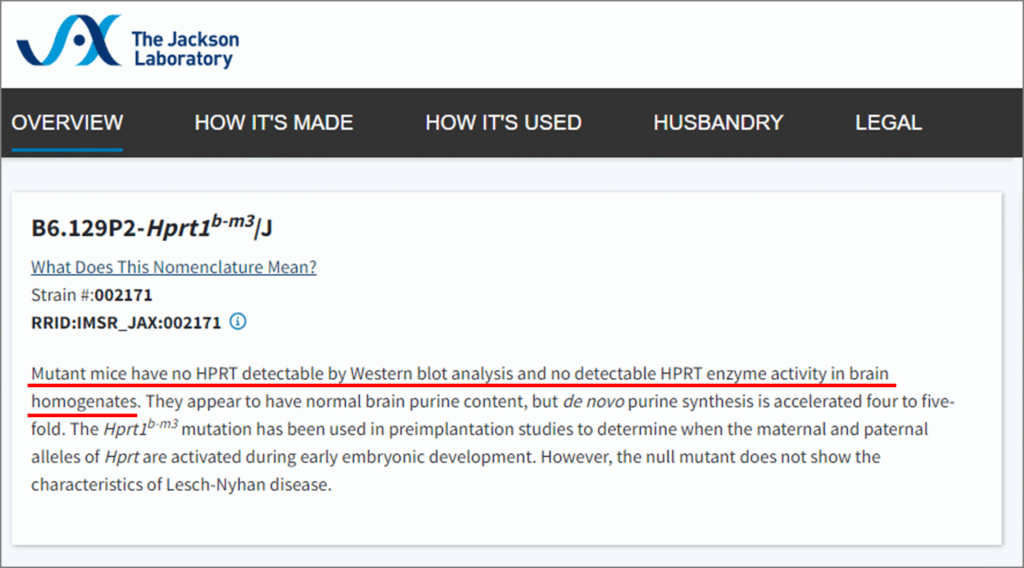
Jackson Laboratory はマウス遺伝学研究において世界的に有名な生物医学研究機関です。
遺伝子改変マウスを含む豊富なマウス系統を提供しており、研究に必要なマウス系統を入手できる可能性があります。
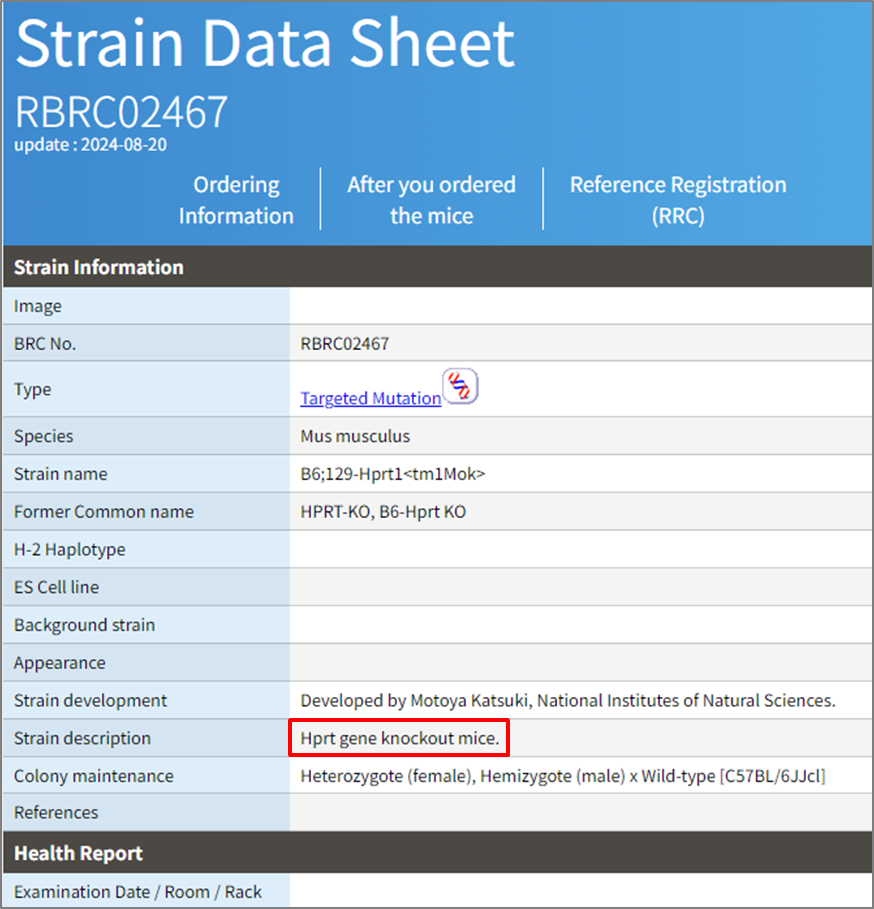
理化学研究所 バイオリソース研究センター(理研BRC)は生命科学研究のための重要な生物資源を提供する国内の主要施設です。マウス、実験植物、細胞材料、遺伝子材料、微生物材料など、多様なバイオリソースを取り扱っています。
理研BRCからマウス系統を入手する場合は、提供されているマウス系統をウェブサイトで検索してみましょう。
その他にも、KOマウスを提供している業者は数多く存在します。目的のKOマウスが購入可能かどうか、幅広く調査してみることをお勧めします。
いいえ:標的遺伝子は Konezumi に登録されているか?
KOマウスの入手が難しい場合など、自身でKOマウスをデザイン・作出する場合には、まずは標的遺伝子がKOnezumiに登録されているかを確認しましょう。
はい:Konezumiが提案するガイドRNAを使用
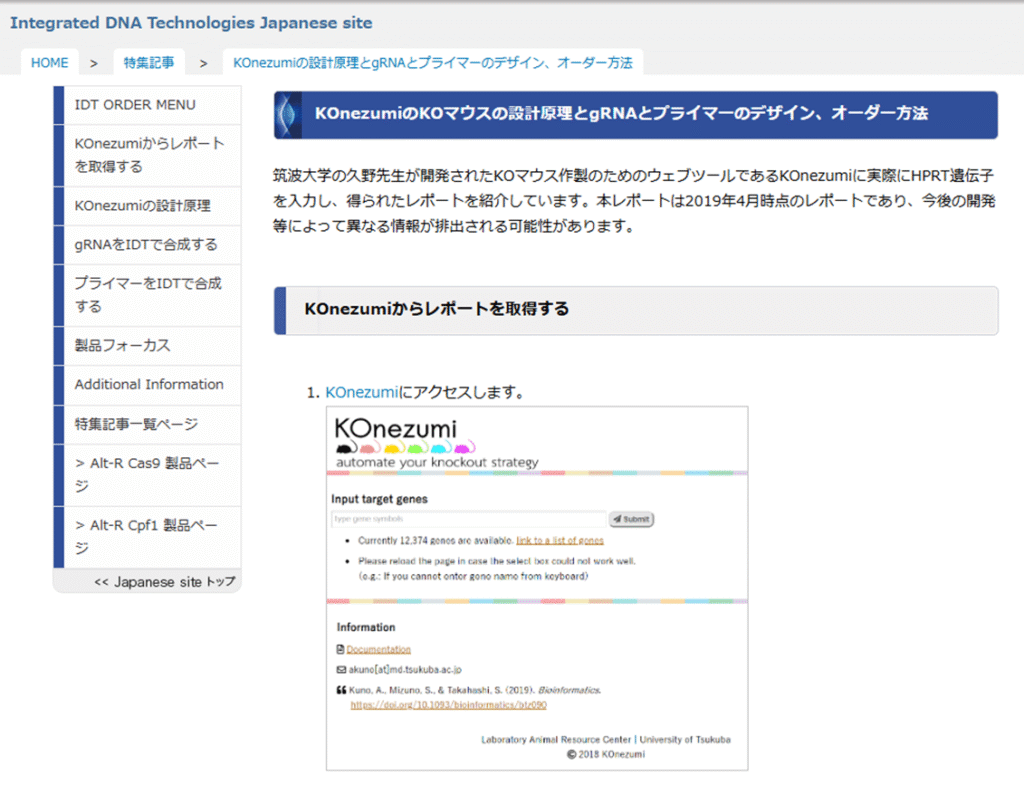
KOnezumi(コネズミ)はKOマウス作製のためのゲノム編集デザインを自動化するWebツールです。筑波大学の研究グループによって開発されました。
KOしたい遺伝子名を入力するだけで、必要となるガイドRNAやジェノタイピング用PCRプライマーを提案してくれます。
マウスの全遺伝子に対応しているわけではありませんが、目的の遺伝子が登録されていれば、研究効率を大幅に向上させることができます。
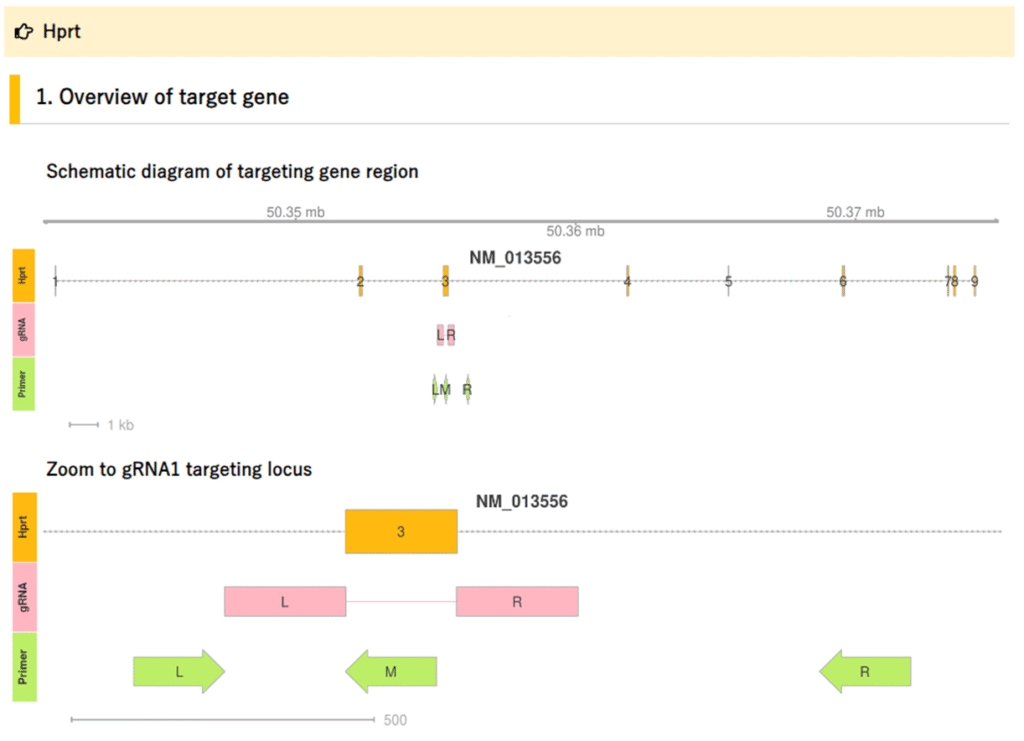
Konezumiは、設計すべきガイドRNAやジェノタイピング用プライマーの位置を視覚的に分かりやすく表示してくれます👇
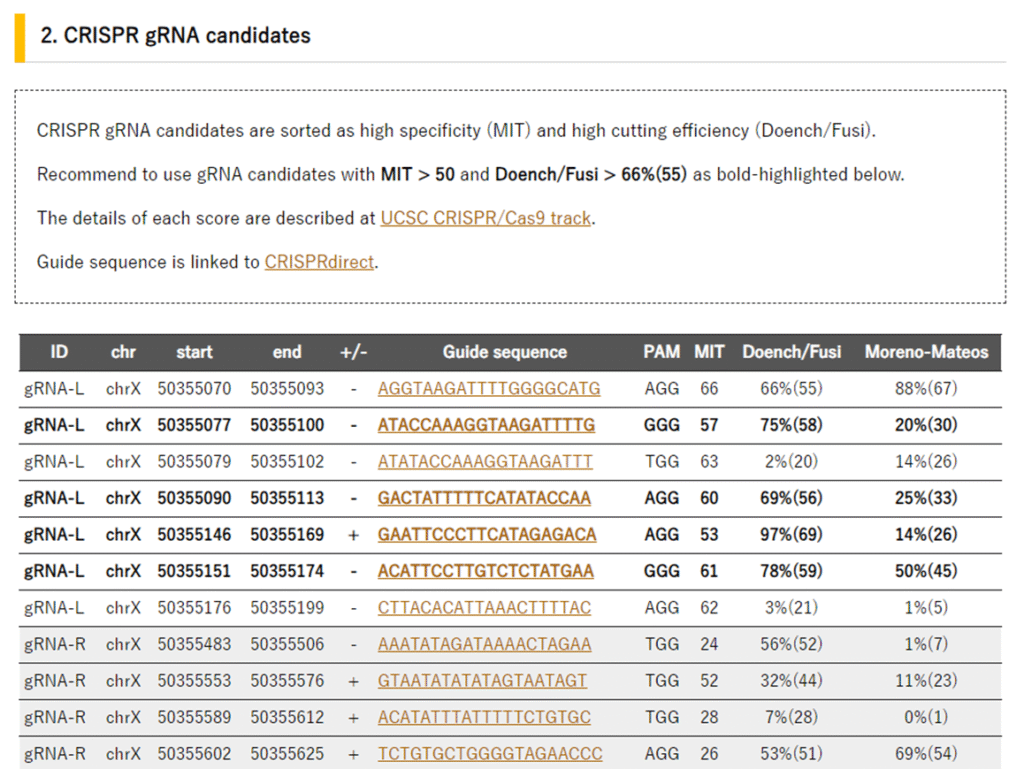
もちろん、ガイドRNAの具体的な配列も詳細に提示してくれます👇
IDTのホームページには、Konezumiの使い方や、IDTを通じてガイドRNAやプライマーを注文する方法が詳しく解説されています。
いいえ:既報と同じKOデザインを使用するか? 欠損するとフレームシフトを起こすエキソンはあるか?
はい:既報のデザインを踏襲、フレームシフトを起こすエキソンを欠損
先行研究のKOマウスデザインを既に把握している場合は、そのデザインを踏襲してKOマウスを作製するのも一つの方法です。
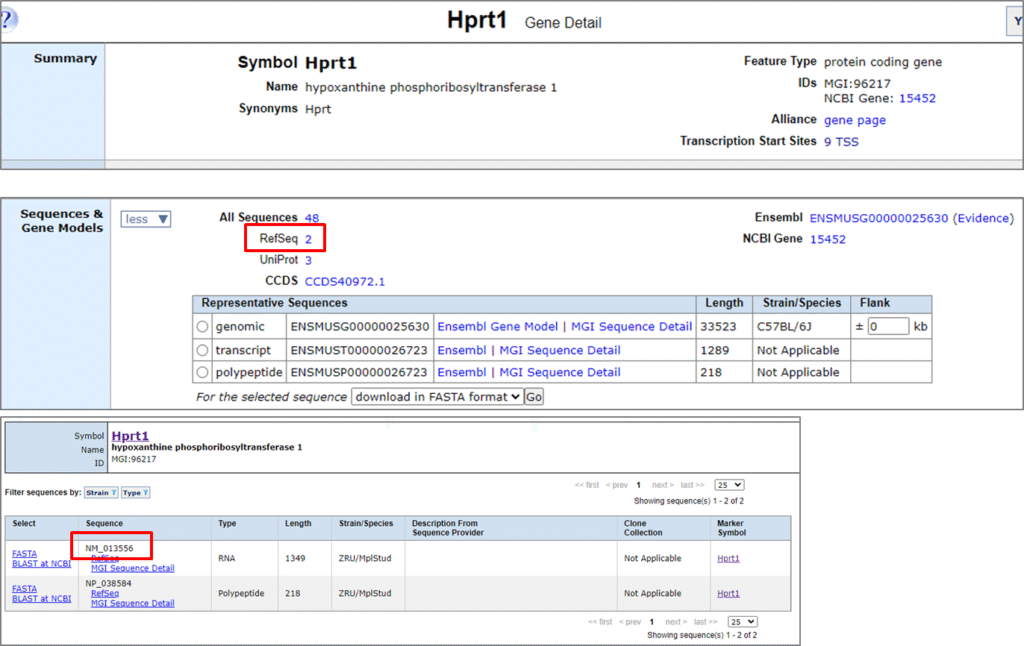
自身でデザインする場合は、配列情報の入手が不可欠です。ここでは、MGIから配列情報を入手し、SnapGeneViewerで確認する方法を紹介します。
MGIでHprt1を検索し、RefSeqからシークエンス番号を確認します。
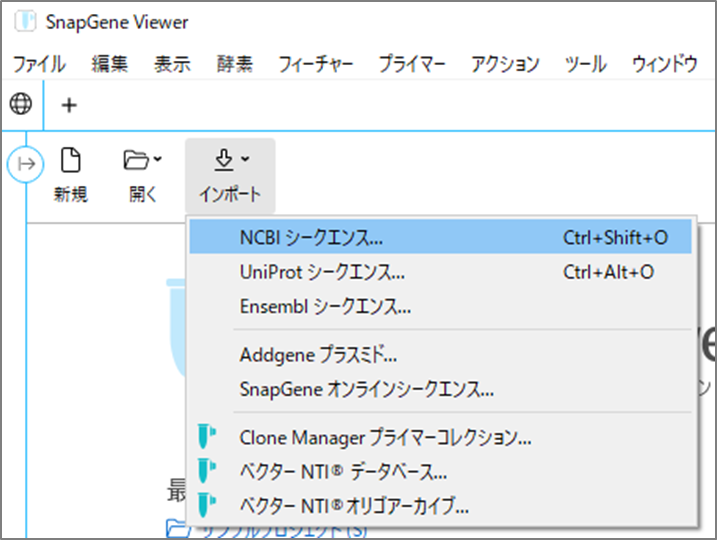
SnapGeneViewerにシークエンス番号を入力します
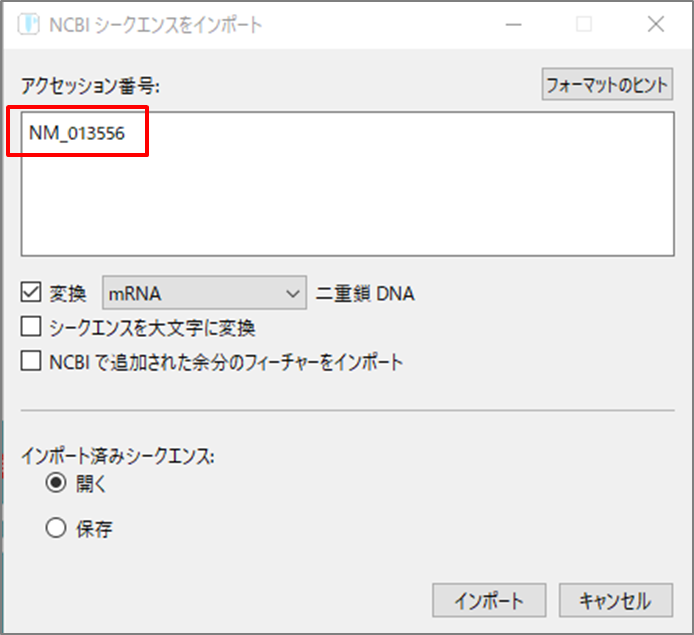
遺伝子の構造や配列を詳細に確認することができます。
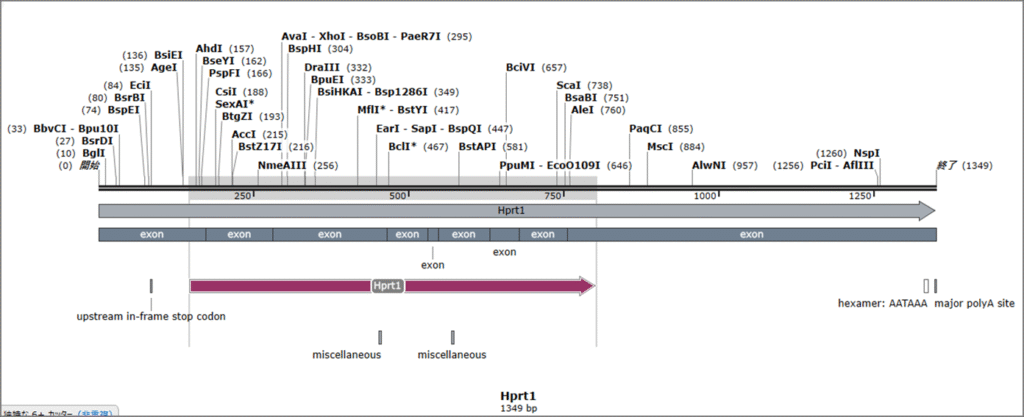
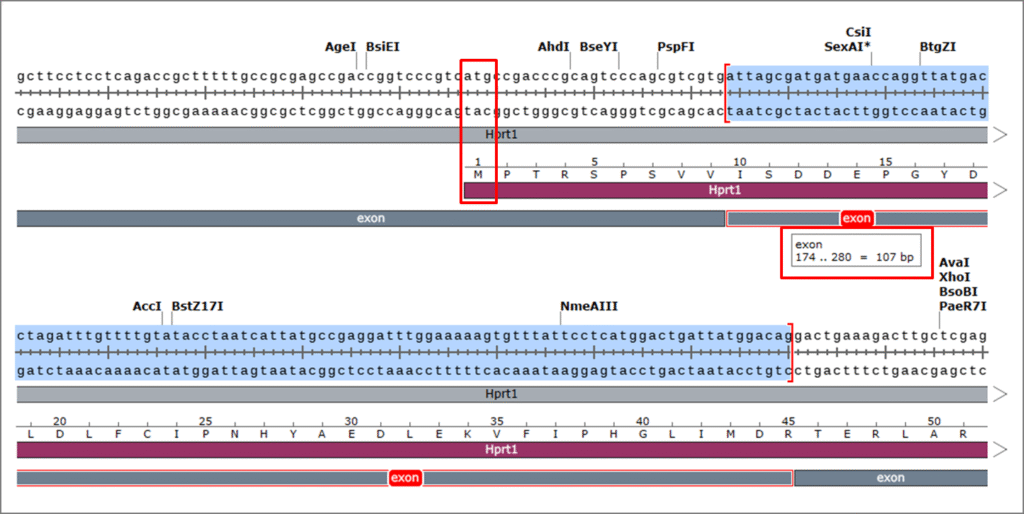
標的エキソンの選定
Hprt1の場合、2番目のコーディングエキソンが107bp(3の倍数でない)となっています。そのため、このエキソンを欠損させることでフレームシフトを起こすことができます。
フレームシフトが起こった場合にできる新しいストップコドンの位置は、事前に精査することが重要です。
エキソンを欠損させてKOを行う場合、1番目のコーディングエキソンを標的とすることはあまりない印象があります。これは、エキソン2以降のATGが新たな開始コドンとして機能する可能性があるためかもしれません。
どのエキソンを標的とすべきかについて、IMPCでは以下の基準を挙げています。
- すべてのトランスクリプトバリアントに含まれること:特定のスプライシングバリアントにのみ含まれるエキソンを欠損させても、他のスプライシングバリアントが機能する可能性があるためです。
- フレームシフトミューテーションを起こすエキソンであること:つまり、3の倍数でない長さのエキソンを選定することが重要です。
- オリジナルのコーディング領域の半分以上が異なる配列になること:例えば、最後のコーディング領域のエキソンを欠損させた場合、元の配列とあまり変わらないアミノ酸配列ができてしまう可能性があります。そのため、できるだけ前方のエキソンを標的とすることが望ましいでしょう。
加えて、フレームシフトの結果、ストップコドンが早期に形成され、できるだけ短いアミノ酸配列が生成されることが望ましいと思います。たとえ元の配列と異なるアミノ酸が生成されても、配列があまりに長いと、それ自体が予期しない機能を持つ可能性があるからです。
ガイドRNAの設計
標的エキソンが決定したら、標的エキソン周辺のゲノム配列(主にイントロン領域)からガイドRNAの候補配列を探索します。ゲノム配列は、先に紹介したMGIなどから入手できます。
ガイドRNAの設計には、CRISPRdirectなど複数のツールが利用可能です。個人的には、各ツールにそこまで大きな差はないと感じるので、使いやすいものを選んで問題ないと思います。
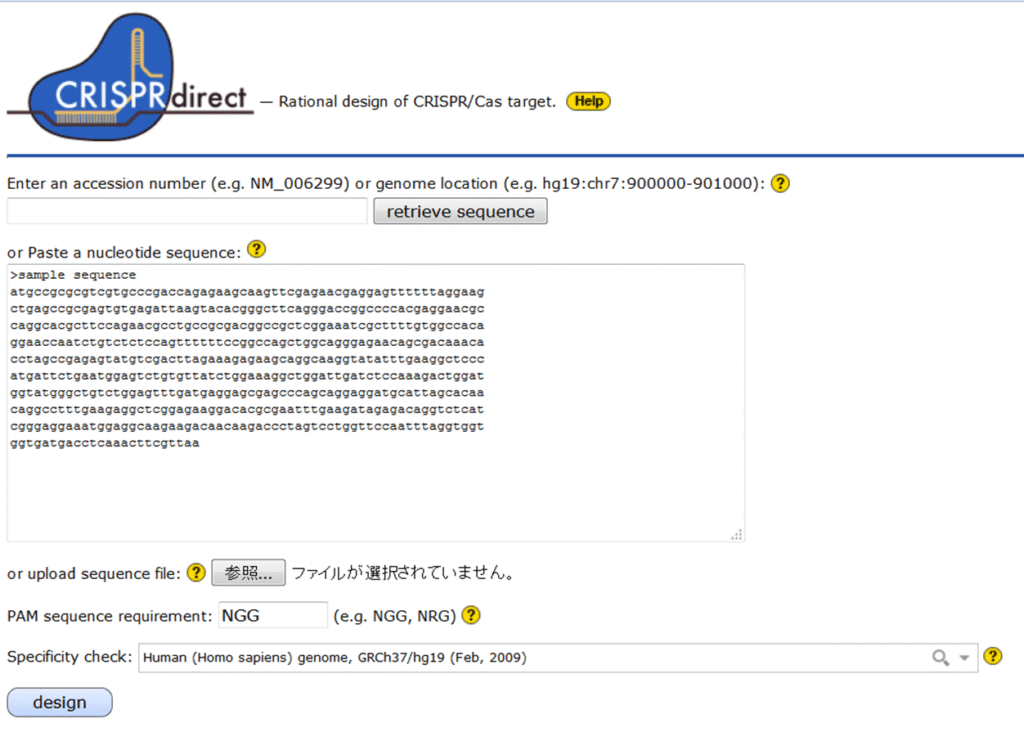
いいえ:遺伝子全領域の欠失、機能欠失、インデルでのフレームシフトを狙う
条件を満たす標的エキソンが見つからず、適切なKO方法が不明な場合の選択肢として、以下の方法を紹介します。
遺伝子全領域の欠失
遺伝子全体、または大部分を欠損させる方法です。遺伝子領域の大半を欠失させることで、遺伝子機能を失わせるアプローチです。
遺伝子領域が長い場合、数 kbpから数十 kbpの欠損になることもありますが、CRISPR/Cas9を用いれば、技術的な難易度はそれほど高くありません。
機能ドメインを標的としたアプローチ
標的遺伝子の重要な機能ドメインが特定されている場合、そこを欠失させるようにガイドを設計する方法も考えられます。
この方法を選択する際は、その機能ドメインを欠失させることで、本当にその遺伝子の機能を完全に失わせることができるかを慎重に検討する必要があります。他にも重要な機能ドメインが存在し、そこが機能してしまう可能性も考慮する必要があります。
「この領域さえ欠失させれば遺伝子の機能は確実に失われる」という確信がある場合や、ある特定の領域の機能を調べたい場合に選択肢となる方法です。
indel(インデル)によるフレームシフト
ガイドRNAを1つだけ設計し、indelを起こしてフレームシフトを狙う方法です。
具体的には、3の倍数でない長さのindel(3n+1、3n+2)を標的遺伝子のコーディングエキソンに導入し、indel以降にフレームシフトを発生させ、早期にストップコドンが形成されることを狙います。
この方法では、機能ドメインの上流、もしくはコーディング領域のできるだけ上流にガイドを設計することが重要です。
また、3n+1または3n+2の各indelが導入された場合に新たに形成されるストップコドンの位置を事前に確認しておきましょう。ストップコドンの位置があまりに下流にあると、indelの結果として作られるタンパク質が長くなり、何らかの機能を保持してしまう可能性があります。
indelを狙う方法の欠点は、どのようなindel(3n、3n+1、3n+2)が発生するかを事前に正確に予測することが困難なことです。indelが発生するかどうかも不確実ですし、発生してもindelの長さが3の倍数でフレームシフトが起きない可能性もあります。
そのため、可能であれば、これまでに紹介したように、ガイドを2つ設計して特定の領域を欠損させるデザインを優先することをおすすめします。
まとめ
ゲノム編集によるKOマウスの作製は、適切な情報収集とガイドRNAの設計が鍵となります。
本記事では、既存リソースの検索方法から、エキソン選定のポイント、具体的な設計支援ツールの活用までを解説しました。
これらの知識を活用することで、効率的かつ失敗の少ないKOマウス作製が可能になります。研究計画の精度向上にぜひお役立てください。








コメント